摘要:
拿到两个数据表,该如何比较这两个表中的元素的相关性呢?
目的:
我手上有两个基因表达矩阵,一个是差异基因的表达矩阵A,另一个是M药靶基因表达矩阵B,两组矩阵如下所示。我现在想绘制对这两个矩阵中的每个基因计算他们之间的相关性,并作图。
| Gene1 | Gene2 | ... | GeneN | |
| Sample1 | ||||
| Sample2 | ||||
| ... | ||||
| SampleM |
思路:
逐列比较两个矩阵(即拿每个矩阵A的列跟每个矩阵B的列进行相关性计算),然后将计算结果整合到新的数据框中,再作图。
步骤:
#创建一个新数据框,用于存放相关性和p值
cor.tar.deg <- data.frame(
Var1 = 'a',
Var2 = 'a',
cor = 0,
p = 0
)
#M.target.exp是M药的靶点的表达矩阵,M.cor.mat.sorted是差异基因的表达矩阵
#逐列计算相关性和显著性
for (i in colnames(M.target.exp)) {
for (j in colnames(M.cor.mat.sorted)) {
cor.res <- cor.test(M.target.exp[[i]], M.cor.mat.sorted[[j]], method='pearson')
cor.tar.deg <- rbind(cor.tar.deg, data.frame(Var1=i, Var2=j, cor=cor.res$estimate, p=cor.res$p.value))
}
}
#因为创建空数据框时第一行留了一个,因此这里把第一行删掉
cor.tar.deg <- cor.tar.deg[-1,]
#将p值用*表示
cor.tar.deg$Signif <- ifelse(cor.tar.deg$p<0.0001, '****',ifelse(cor.tar.deg$p<0.001, '***', ifelse(cor.tar.deg$p<0.01, '**', ifelse(cor.tar.deg$p<0.05, '*', ''))))
#我们使用ggplot来可视化(尽管pheatmap做热图更简单,但ggplot相比之下更加灵活)
ggplot(cor.tar.deg, aes(x=Var1,y=factor(Var2, levels = cor.tar.deg$Var2[1:84]), fill=cor, label=Signif)) + theme_classic() + #这里我希望我的基因顺序按照它们之前在M.cor.mat.sorted矩阵中的顺序来,因此用factor处理了一下。如果这边不处理,ggplot会默认按照字母顺序来排序。
geom_tile() +
scale_fill_gradient2(low = '#91d1c2ff', mid = 'white', high = '#DC0000ff') +
geom_text(aes(label=Signif)) +
ylab('DEGs of preR vs PreS') + xlab('Midostaurin targets') +
theme(axis.line.x = element_blank(), axis.line.y = element_blank(), axis.ticks.x = element_blank(), axis.ticks.y = element_blank())
以上代码执行完成后,我们将会得到一个类似这样的热图:
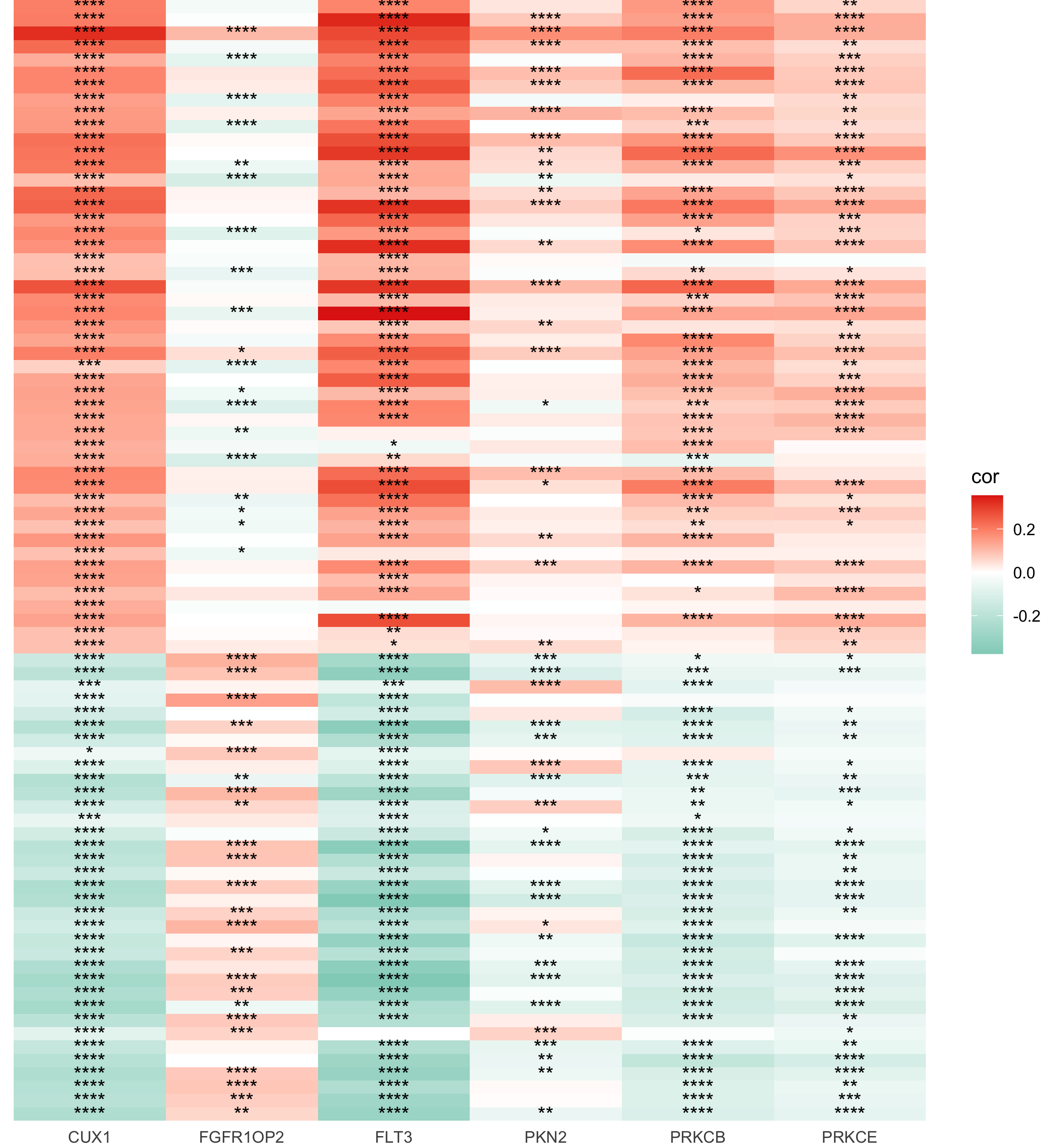
大功告成!
最后:
其实很多封装好的包,例如pheatmap,corrplot等等都能做类似的图。如果你对你的图没啥特殊的要求,它们完全能满足日常需求。但当你想在你的图里尽可能的多添加一些信息时,或者希望美化你的图,那这些包往往都是捉襟见肘。总的来说,只要懂基本的作图逻辑,那ggplot几乎可以做出一切你想要的图。这也是为什么我现在逐渐脱离那些高度封装的作图包而研究ggplot复杂作图的主要原因。
 苏公网安备 32050602011302号
苏公网安备 32050602011302号